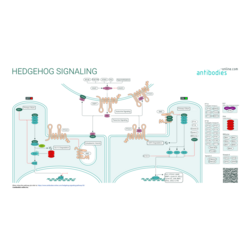
Signalisation Hedgehog
Le signal Hedgehog (Hh) se conserve parfaitement à travers les cordés de différentes classes taxonomiques. Il est essentiel dans le développement de l’embryon. La voie Hedehog a été décrite et caractérisée pour la première fois chez la mouche du vinaigre, la Drosophila melanogaster. Chez les mammifères, le signal Hh régule le sort des cellules, la polarité des tissus, et la structure cellulaire durant l’embryogénèse précoce et la morphogénèse de certains organes et de certains tissus. Par conséquent, il est mis en latence dans la plupart des tissus adultes, mais il peut être réactivé suite à des blessures afin de stimuler la réparation et la régénération.
Différents ligands Hh, dont les homologues sonic hedgehog (SHH), indian hedgehog (IHH), et desert hedgehog (DHH) sont produits en tant que précurseurs qui soutiennent le clivage autocatalytique, l’attachement du cholestérol C-terminal, et la palmitylation N-terminale avant la sécrétion. La libération et l’accumulation extracellulaire des ligands matures sont régulées par les homologues de la protéine dispatchée (Dips) de la Drosophile. La liaison entre les ligands Hh sécrétés et l’homologue récepteur (PTCH1, PTCH2) déclenche la stimulation du réseau de signaux : l’effet inhibiteur de PTCH sur le récepteur transmembranaire SMO est supprimé, ce qui conduit à l’activation des facteurs de transcription de l’oncogène lié au gliome (GLI).
Une structure-clé spécialisée de ce processus est le cil primaire en microtubule. En l’absence du Hh non traité, les protéines GLI non activantes, ainsi que leur régulateur SUFU, sont concentrées à la pointe distale du cil primaire. À partir de la liaison du ligand Hh, PTCH se relocalise au niveau de la surface cellulaire, rendant ainsi possible la translocation du SMO vers le cil primaire et la régulation à la baisse du SUFU. Plusieurs protéines du cil primaire, essentielles du point de vue de la structure, ont également des effets régulateurs sur la cascade de signaux Hh.
Une régulation aberrante de Hh/GLI entraîne des problèmes graves pour les tissus et l’apparition de nombreux cancers agressifs. La cascade Hh/GLI a également été mise en relation avec la régulation des gènes déterminant le caractère souche des cellules et avec la survie des cellules-souches des cancers.
References
- : "Hedgehog Pathway Inhibitors: A New Therapeutic Class for the Treatment of Acute Myeloid Leukemia." dans: Blood cancer discovery, Vol. 1, Issue 2, pp. 134-145, (2022) (PubMed).
- : "The emerging conceptual framework of evolutionary developmental biology." dans: Nature, Vol. 415, Issue 6873, pp. 757-64, (2002) (PubMed).
- : "Posttranscriptional regulation of smoothened is part of a self-correcting mechanism in the Hedgehog signaling system." dans: Molecular cell, Vol. 6, Issue 2, pp. 457-65, (2000) (PubMed).
- : "Anti-patched-1 antibodies suppress hedgehog signaling pathway and pancreatic cancer proliferation." dans: Anticancer research, Vol. 27, Issue 6A, pp. 3743-7, (2007) (PubMed).
- : "The hedgehog signaling pathway: where did it come from?" dans: PLoS biology, Vol. 7, Issue 6, pp. e1000146, (2009) (PubMed).
- : "The Hedgehog protein family." dans: Genome biology, Vol. 9, Issue 11, pp. 241, (2009) (PubMed).
- : "Transducing the hedgehog signal." dans: Cell, Vol. 103, Issue 3, pp. 371-4, (2000) (PubMed).
- : "The hedgehog signaling network." dans: American journal of medical genetics. Part A, Vol. 123A, Issue 1, pp. 5-28, (2004) (PubMed).