CUT&RUN
CUT&RUN est l'acronyme de "cleavage under targets and release using nuclease" (clivage sous les cibles et libération à l'aide de nucléases). La technique développée par le laboratoire Henikoff offre une nouvelle approche de l'épigénétique. CUT&RUN introduit quelques modifications majeures pour éliminer les défauts inhérents à ChIP-seq. Elle est simple à réaliser et intrinsèquement robuste, avec un bruit de fond extrêmement faible ne nécessitant qu'un dixième de la profondeur de séquençage de la technique ChIP. CUT&RUN est rentable pour le profilage des facteurs de transcription et de la chromatine. Vous ne connaissez pas encore le CUT&RUN ? Découvrez l'enregistrement et le poster de notre webinaire d'introduction pour en savoir plus sur cette application !
Vous êtes nouveau sur CUT&RUN ? Découvrez notre introduction webinaire et poster pour en savoir plus sur cette application !
CUT&RUN Antibodies
Une gamme d'anticorps validés pour une utilisation dans les tests CUT&RUN.
Sets CUT&RUN
CUT&RUN en toute simplicité - avec nos sets et composants
CUT&RUN Perles Concanavalin A
Perles magnétiques ConA pour utilisation dans les essais CUT&RUN
CUT&RUN - Mieux que ChIP-seq!
CUT&RUN Workflow: Extraction sans fragmentation
CUT&RUN est réalisé in situ sur des cellules immobilisées et intactes sans réticulation. L'ADN est obtenue en utilisant la nucléase micrococale fusionnée à la protéine A et/ou à la protéine G (pAG-MNase). G (pAG-MNase). La protéine de fusion est dirigée vers la cible souhaitée par la liaison de la fraction de la protéine A/G à la protéine G. the Protein A/G moiety to the Fc region of an antibody bound to the target. DNA under the target is subsequently cleaved and released and the pAG-MNase-antibody-chromatin est libre de diffuser hors de la cellule. DNA cleavage products are extracted and then traités par séquençage de nouvelle génération (NGS).
Le protocole standard CUT&RUN est principalement destiné au profilage des protéines histones et des facteurs de transcription qui se lient à la chromatine. facteurs de transcription qui se lient à la chromatine. Détection fiable des facteurs de transcription rares, d'interactions transitoires ou de protéines dans des complexes qui ne sont pas directement associés à l'ADN. ADN s'est avérée difficile en utilisant CUT&RUN sur des échantillons non réticulés. Préservation de la protéine-protéine protéine-protéine et ADN-protéine en utilisant des réticulants peut introduire des artefacts. artefacts. La représentation suivante montre le protocole standard de CUT&RUN :
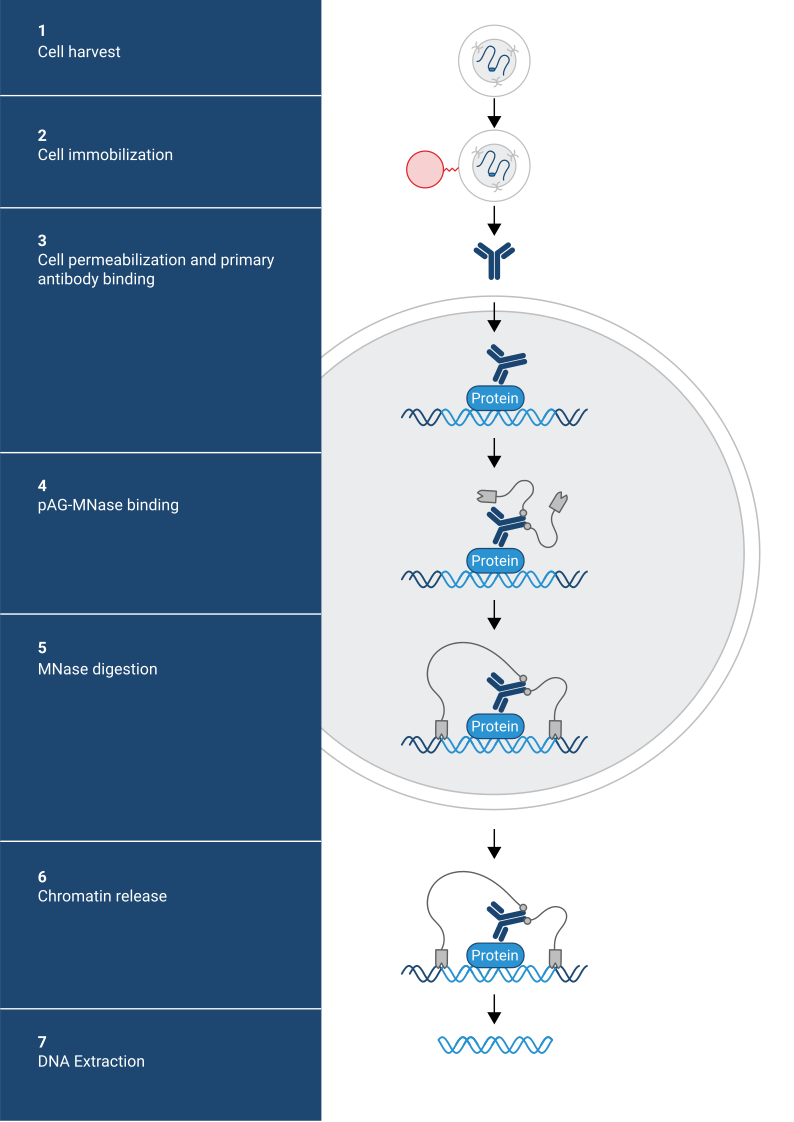
Immobilisation et perméabilisation des cellules Step, Liaison de l'anticorps primaire CUT&RUN, Liaison et digestion de la pAG-MNase, Libération de la chromatine
CUT&RUN Low Volume and Urea (CUT&RUN Lov-U) is a modification of the original procedure aimed at difficult to characterize interactions in situ while avoiding crosslinking. Initial nuclear extraction minimizes sequestration of the primary antibody and pAG-MNase by non-not chromatin-bound targets in the cytosol. Low volumes throughout the protocol facilitate parallel processing of samples which benefits scalability and reprodcibility. Lastly, urea is used as chaotropic reagent to denature samples in situ and release CUT&RUN products, followed by DNA clean-up on beads.
L'un des inconvénients de CUT&RUN est la nécessité de polir et de ligaturer l'adaptateur de séquençage avant la préparation de l'échantillon. nécessité de polir et de ligaturer l'adaptateur de séquençage avant la préparation d'une d'une bibliothèque de séquençage. Une combinaison du protocole CUT&RUN et de la ligature par un adaptateur de séquençage "hyperactif" a été mise au point. hyperactif transposase Tn5 hyperactive a donné naissance à la méthode CUT&Tag. Les cellules sont immobilisées à l'aide de billes magnétiques de Concanavalin A et perméabilisées de manière réversible à l'aide de digitonine. en utilisant de la digitonine. Cependant, au lieu de la coupure dirigée par une nucléase, l'ADN est fragmenté par un pA/G-Tn5 chargé de duplex d'adaptateurs de séquençage. Les adaptateurs de séquençage sont attachés aux fragments d'ADN directement pendant le marquage. Aucun autre traitement final de l'ADN est d'ADN n'est nécessaire et les fragments peuvent être utilisés pour la préparation de bibliothèques de séquençage. En outre, CUT&Tag est intrinsèquement moins sensible aux dommages endogènes de l'ADN que CUT&RUN, car la transposition a lieu sur des molécules d'ADN double brin intactes. la transposition a lieu sur des molécules d'ADN double brin intactes.
En savoir plus sur CUT&Tag : Flux de travail, avantages, produits
CUT&RUN Antibodies
A range of antibodies validated for use in CUT&RUN assays.
CUT&RUN Sets
CUT&RUN made easy - with our sets & components
CUT&RUN ConA Beads
Magnetic ConA Beads for usage in CUT&RUN assays
Discover our CUT&RUN Antibodies!
Show moreAntigènes cibles pour CUT&RUN et CUT&Tag
Les modifications des histones, telles que H3K27me3 (répressive) ou H3K4me3 (associée aux promoteurs actifs), sont couramment utilisées comme antigènes dans les techniques CUT&RUN et CUT&Tag. D’autres antigènes incluent des régulateurs de la transcription comme les facteurs de transcription CTCF ou SOX2, des modificateurs de la chromatine comme HDAC2, ou encore des protéines impliquées dans le métabolisme des acides nucléiques, telles que Pol II. Les G-quadruplexes et la m6A font partie des cibles non protéiques. Des étiquettes comme FLAG, HA ou GFP sont des options pour les protéines cibles exprimées de manière recombinante.

