Épigénétique
L'épigénétique se définit comme des changements héréditaires de l'expression des gènes qui, contrairement aux mutations, ne sont pas attribuables à des altérations de la séquence de l'ADN. Les mécanismes épigénétiques prédominants sont la méthylation de l'ADN, les modifications de la chromatine, la perte d'empreinte et les ARN non codants. La régulation épigénétique de l'expression des gènes semble avoir des effets à long terme et des effets de grande ampleur sur la santé.1 Il existe une implication claire et prémonitoire des avantages pour la recherche en science fondamentale et en santé humaine qui découlent d'une compréhension plus complète de l'épigénome. Ces avantages ont poussé les chercheurs à affluer dans ce domaine, et les organismes de financement ont suivi le mouvement.
Une grande partie de l'épigénétique est contrôlée par les modifications post-traductionnelles sur les protéines. Les méthylations, les acétylations, les ubiquitinations ou les phosphorylations entraînent des modifications qui influencent par exemple la transcription. Les modifications épigénétiques se produisent souvent au cours de la vie d'un organisme ; toutefois, ces changements peuvent être transférés à la génération suivante s'ils se produisent dans les cellules germinales. La paramutation, la mise en signet, l'empreinte, le silenciement des gènes, l'inactivation du chromosome X, l'effet de position, le trouble modifiable ou la sévérité phénotypique, la reprogrammation, les attributs maternels, les processus cancérigènes, les effets tératogènes, la régulation des modifications des histones, les états de l'hétérochromatine et le clonage sont connus pour impliquer des processus épigénétiques.2
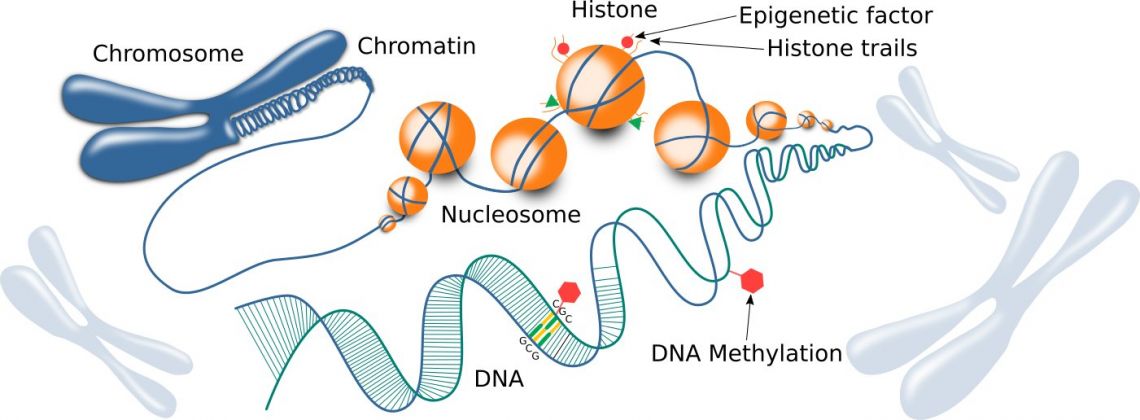
Fig. 1 : Structure de la chromatine, y compris les histones et l'ADN.
Modifications de l'ADN
La fixation covalente d'un groupe méthyle à la position C5 de la cytosine comprend la principale modification épigénétique de l'ADN. La présence d'une cytosine méthylée peut réprimer la transcription en inhibant la liaison des facteurs de transcription ou peut favoriser la liaison d'autres répresseurs de transcription, y compris les protéines modificatrices d'histones, telles que les histones désacétylases.3
La déméthylation de l'ADN joue un rôle important dans le développement et la tumorigenèse chez les mammifères. La déméthylation de l'ADN, qui se produit dans les cellules germinales primordiales et dans les embryons précoces, est essentielle pour que les cellules reviennent à un état pluripotent.4
Modifications des histones
Similairement à la méthylation de l'ADN, les modifications post-traductionnelles des histones n'affectent pas la séquence nucléotidique de l'ADN mais peuvent modifier sa disponibilité pour la machinerie transcriptionnelle. Plusieurs types de modifications des histones sont connus, parmi lesquels l'acétylation, la méthylation, la phosphorylation et l'ubiquitination sont les plus étudiés et les plus importants en termes de régulation de la structure et de l'activité (transcriptionnelle) de la chromatine. 5 Alors que la méthylation de l'ADN est considérée comme une modification épigénétique très stable, les modifications des histones sont comparativement plus labiles, leurs niveaux étant maintenus par un équilibre entre les enzymes de modification des histones, qui ajoutent ou suppriment des modifications spécifiques. Les niveaux aberrants de modification des histones résultent d'un déséquilibre de ces enzymes modificatrices dans les tissus malades, ainsi la correction du niveau accru ou diminué d'une enzyme particulière rétablira l'équilibre naturel dans les cellules affectées.6
Remodelage de la chromatine
La présence d'histones agit comme une barrière à l'accès des protéines ; ainsi, le remodelage de la chromatine doit se produire pour les processus essentiels tels que la transcription et la réplication. En conjonction avec les modifications des histones, la méthylation de l'ADN joue des rôles critiques dans l'extinction des gènes par le remodelage de la chromatine. Le remodelage de la chromatine est également lié à la réponse aux dommages causés par l'ADN, au maintien des propriétés des cellules souches et aux programmes de différenciation cellulaire. Il est de plus en plus évident que les modifications de la chromatine produisent des altérations durables de la structure de la chromatine et de la transcription. Des études récentes ont montré que les expositions environnementales in utero ont le potentiel de modifier les réseaux de signalisation normaux du développement, les réponses physiologiques et la susceptibilité aux maladies plus tard dans la vie au cours d'un processus connu sous le nom de reprogrammation développementale.7
Facteurs de transcription
Les modifications chimiques des histones et de l'ADN, telles que la méthylation des histones, l'acétylation des histones et la méthylation de l'ADN, jouent des rôles critiques dans la régulation épigénétique des gènes. De nombreuses enzymes qui ajoutent ou suppriment ces modifications chimiques sont connues, ou peuvent être soupçonnées, d'être sensibles aux changements du métabolisme intracellulaire. Elles utilisent (souvent) des cosubstrats générés par le métabolisme cellulaire, fournissant ainsi un lien potentiel entre la nutrition, le métabolisme et la régulation des gènes.8
Anticorps populaires pour l'épigénétique
- (1)
- (9)
- (1)
- (8)
- (14)
- (1)
- (7)
- (20)
- (1)
- (4)
- (21)
- (1)
- (4)
- (1)
- (4)
- (5)
- (1)
- (3)
- (5)
- (1)
- (1)
- (1)
- (1)
- (3)
Vous avez besoin d'aide ? Appelez notre support client PhD!
- Nous vous aidons à trouver le bon produit pour votre recherche.
- Nous offrons des anticorps, des kits, des protéines, des lysats fiables pour votre recherche.
- Contactez-nous par courriel ou par téléphone : (877) 302 8632 (US) ou +49 241 95 163 153 (International) .
Informations et produits connexes
Introduction à l'immunoprécipitation de la chromatine (ChIP)
CUT&RUN - Méthode améliorée d'étude des interactions protéine-ADN
References
- : "Epigenetics: principles and practice." dans: Digestive diseases (Basel, Switzerland), Vol. 29, Issue 2, pp. 130-5, (2011) (PubMed).
- : "Role of Epigenetics in Biology and Human Diseases." dans: Iranian biomedical journal, Vol. 20, Issue 5, pp. 246-58, (2017) (PubMed).
- : "Epigenetic modifications: basic mechanisms and role in cardiovascular disease (2013 Grover Conference series)." dans: Pulmonary circulation, Vol. 4, Issue 2, pp. 169-74, (2014) (PubMed).
- : "DNA methylation: superior or subordinate in the epigenetic hierarchy?" dans: Genes & cancer, Vol. 2, Issue 6, pp. 607-17, (2011) (PubMed).
- : "Histone modifications and their role in epigenetics of atopy and allergic diseases." dans: Allergy, asthma, and clinical immunology : official journal of the Canadian Society of Allergy and Clinical Immunology, Vol. 14, pp. 39, (2018) (PubMed).
- : "Epigenetic modifications as therapeutic targets." dans: Nature biotechnology, Vol. 28, Issue 10, pp. 1069-78, (2015) (PubMed).
- : "Long-term effects of chromatin remodeling and DNA damage in stem cells induced by environmental and dietary agents." dans: Journal of environmental pathology, toxicology and oncology : official organ of the International Society for Environmental Toxicology and Cancer, Vol. 32, Issue 4, pp. 307-27, (2014) (PubMed).
- : "Influence of metabolism on epigenetics and disease." dans: Cell, Vol. 153, Issue 1, pp. 56-69, (2013) (PubMed).




