Protéines SARS-CoV-2
anticorps-enligne fournit une large sélection de protéines recombinantes pour la recherche sur le SARS-CoV-2, notamment protéine membranaire, protéine nucléocapside, protéine de pointe, protéine S1 mutations, protéine d'enveloppe, et protéines non structurelles. Découvrez notre portefeuille de produits.
- Incl. N501Y Mutation.
- Source: HEK-293 Cells.
- Pour les tests ELISA et Ligand Binding Assay.
- Livraison en 2 à 3 jours.
- T19R, L452R, T478K, D614G, P681R, D950N
- Source: HEK-293 Cells
- Rho-1D4 Tag.
- Livraison en 3 à 4 jours.
- Trimérique, protéine de pointe Cov-2 pleine longueur.
- Pour le développement de tests.
- Livraison en 3 à 4 jours.
- Protéine Spike S1 étiquetée avec His-tag.
- Pour les études fonctionnelles.
- Numéro de produit ABIN6952427

Produits supérieurs : Protéine de pointe du SRAS-CoV-2 trimérique
- (2)
- (6)
- (5)
- (5)
- (4)
- (4)
- (4)
*Super Stable Trimer: Des substitutions de proline (F817P, A892P, A899P, A942P, K986P, V987P) et d'alanine (R683A et R685A) sont introduites pour stabiliser l'état de préfusion trimérique de la protéine SARS-CoV-2 S et supprimer le site de clivage de la furine, respectivement.
Sommet du SARS-CoV-2 (S1, S2) Protéine
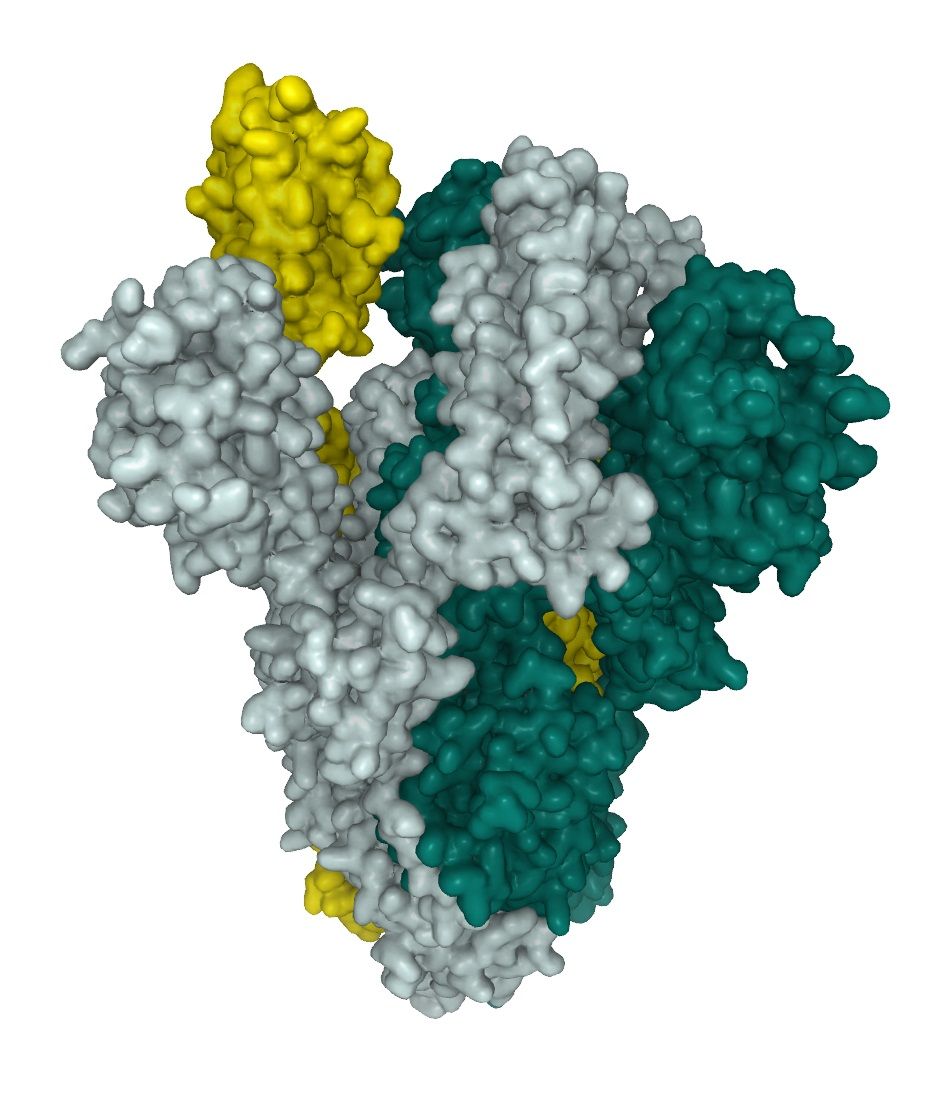
CoV utilise sa glycoprotéine de pointe (S), une cible principale pour l'anticorps de neutralisation, pour lier son récepteur, et assurer la fusion de la membrane et l'entrée du virus. Chaque monomère de la protéine S trimérique est d'environ 180 kDa, et contient deux sous-unités, S1 et S2, qui servent respectivement de médiateur pour la fixation et la fusion membranaire.(6)
- (9)
- (6)
- (5)
- (5)
- (4)
- (5)
- (4)
- (3)
- (3)
- (2)
- (2)
- (2)
- (2)
Nucléocapside (N) du SARS-CoV-2 Protéine
La protéine de la nucléocapside est une protéine de structure importante pour les coronavirus. Elle est très abondante dans les virus. Sa fonction consiste à pénétrer dans la cellule hôte, à se lier au génome de l'ARN viral et à former le noyau de la ribonucléoprotéine.(7) La protéine N contient deux domaines distincts de liaison à l'ARN (NTD et CTD) liés par une région de liaison mal structurée contenant un domaine riche en sérine/arginine (SR).
- (2)
- (5)
- (2)
- (1)
- (2)
- (1)
- (1)
- (1)
- (1)
- (1)
- (2)
- (2)
- (2)
- (2)
- (2)
- (2)
- (2)
Membrane (M) du SARS-CoV-2 Protéine
La protéine de la membrane du coronavirus (M) est l'acteur clé de l'assemblage du virion. L'une de ses fonctions est de servir de médiateur pour l'incorporation des pics dans l'enveloppe virale. Lorsqu'elle est exprimée seule, elle s'accumule dans le complexe de Golgi en complexes homomultimériques.
Enveloppe du SARS-CoV-2 (E) Protéine
La protéine E du SARS-CoV-2 est une protéine longue de 75 acides aminés existant sous forme monomérique et homo-pentamérique. Environ 20 copies de la protéine ont été trouvées dans la particule virale et des études antérieures basées sur la mutagenèse ont démontré son rôle essentiel dans l'apparition et le développement de l'infection virale.9
- (1)
- (2)
- (1)
SARS-CoV-2 Protéines non structurelles (NSP)
Le génome du SARS-CoV-2 code 16 protéines non structurelles (Nsp1-16), quatre protéines structurelles et neuf facteurs accessoires supposés.(8) Les NSP comprennent les divers enzymes et facteurs de transcription que le virus utilise pour se répliquer, tels que la protéase virale, la réplicase de l'ARN et les protéines de contrôle de l'hôte.
- (2)
- (1)
- (1)
- (1)
- (1)
- (1)
- (1)
Le rôle des protéines recombinantes dans la recherche sur le SARS-CoV-2
Les protéines recombinantes SARS-CoV-2 sont utilisées comme antigènes pour le développement d'anticorps, comme antigènes de capture ou comme étalons dans les tests. Elles peuvent être utilisées comme contrôle positif dans les ELISA de détection d'antigènes pour séparer avec précision les résultats positifs réels des résultats potentiellement faux ou comme antigènes de capture pour les ELISA d'immunoglobine Le trimère, full length SARS-CoV-2 Spike protein par exemple, est adapté au développement de tests et très utile pour l'étude des anticorps neutralisants. En outre, les protéines SARS-CoV-2 sont nécessaires pour la découverte de médicaments et les études de reconversion des médicaments. Dans le processus de découverte de médicaments, les études fonctionnelles avec des protéines actives sont essentielles pour vérifier les effets inhibiteurs de la substance testée. Les PSN ainsi que les protéines N sont à l'honneur en tant que cibles potentielles ; Leurs fonctions et leur interaction avec la cellule hôte sont cruciales pour la propagation du virus et donc très pertinentes pour les stratégies d'inhibition. Il a été démontré que la protéine N du SARS-CoV-2 affecte le système du complément, tandis que les PSN du SARS-CoV-2 sont responsables de la réplication du virus.
Produits connexes: full length SARS-CoV-2 Spike protein (ABIN6952670)
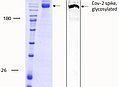
Les modifications post-traductionnelles (PTM), comme la glyscosilation, modifient les protéines en dernière étape de la maturation pour favoriser le repliement des protéines et améliorer leur stabilité. Le schéma de glycosylation de la protéine de pointe du SARS-CoV-2 permet de comprendre la structure du virus et est important pour l'identification des immunogènes pour la conception de vaccins, notamment en ce qui concerne l'encombrement stérique. La glycoprotéine de pointe existe sous la forme d'une protéine de fusion homotrimérique. Chacun des trimères contient 66 sites de glycosylation pour les glycanes N-liés à l'hôte. Dans l'état prédominant du trimère, un des RBD est en position "haute" tandis que les deux autres sont en position "basse". L'interaction entre la protéine S et l'ACE2 n'a lieu qu'avec un seul RBD en position "haute".
Le SARS-CoV-2 utilise une structure à haute teneur en mannose ainsi que des glycanes de type complexe sur leurs protéines de pointe (voir ci-dessous). 1 Cela conduit à une structure de surface complexe, un défi pour la recherche d'interacteurs et la génération d'anticorps neutralisants (Nabs). La Full length SARS-CoV-2 Spike protein (ABIN6952670) est, comme nos autres protéines actives SARS-CoV-2, produite dans les cellules HEK293. Le système d'expression des mammifères est capable d'imiter le schéma de glycosilation complexe du SARS-CoV-2 et d'exprimer la protéine de fusion homotrimérique dans son état actif "up" (fig.1). La Full length SARS-CoV-2 Spike protein est donc très utile pour l'étude des anticorps neutralisants.
Dans un article récent sur la glycobiologie, Shajahan et al. ont réalisé un profilage quantitatif spécifique au site de glycanes liés à l'azote et liés à l'oxygène sur des protéines recombinantes SARS-CoV-2 S subunit S1 et SARS-CoV-2 S protein subunit S2 par glycoprotéomie en utilisant la LC-MS/MS haute résolution. La protéine de pointe est composée de deux sous-unités protéiques (S1 et S2), qui possèdent ensemble 22 sites potentiels de N-glycosylation. Le groupe a identifié 2 sites de O-glycosylation inattendus au niveau du domaine de liaison au récepteur (RBD) de la sous-unité S1.1
Produits connexes: SARS-CoV-2 Spike Subunit S1 (AA 16-690) protein (His tag) (ABIN6952319) | SARS-CoV-2 Spike Subunit S2 (AA 697-1213) protein (His tag) (ABIN6952319)
Les N-glycanes de la protéine S jouent un rôle important dans le repliement correct des protéines et l'amorçage par les protéases de l'hôte. Comme les glycanes peuvent protéger les résidus d'acides aminés et autres épitopes des cellules et de la reconnaissance des anticorps, la glycosylation peut permettre au coronavirus d'échapper aux réponses immunitaires innées et adaptatives. 2,3,4 Le groupe a utilisé des SARS-CoV-2 S1 Protein et SARS-CoV-2 S2 Protein recombinants exprimés dans des cellules HEK293 et a observé une occupation partielle des N-glycanes sur 17 des 22 sites de N-glycosylation. Des chaînes de sucre de type mannose élevé (Man5GlcNAc2) ont été mises en œuvre comme structure prédominante sur tous les sites.5
Thr323 et Ser325 ont été identifiés comme des sites de O-glycosylation sur la sous-unité S1 de la protéine de pointe SARS-CoV-2 par profilage glycoprotéomique par spectrométrie de masse à haute résolution. Les résidus Thr323 et Ser325 sont situés à la RBD de la sous-unité S1 du SARS-CoV-2, et donc la O-glycosylation à cet endroit pourrait jouer un rôle critique dans la liaison virale avec les récepteurs hACE2.3
Références
: "Deducing the N- and O-glycosylation profile of the spike protein of novel coronavirus SARS-CoV-2." dans: Glycobiology, Vol. 30, Issue 12, pp. 981-988, (2020) (PubMed).: "Mapping Neutralizing and Immunodominant Sites on the SARS-CoV-2 Spike Receptor-Binding Domain by Structure-Guided High-Resolution Serology." dans: Cell, Vol. 183, Issue 4, pp. 1024-1042.e21, (2020) (PubMed).
: "The proximal origin of SARS-CoV-2." dans: Nature medicine, Vol. 26, Issue 4, pp. 450-452, (2020) (PubMed).
: "Global aspects of viral glycosylation." dans: Glycobiology, Vol. 28, Issue 7, pp. 443-467, (2018) (PubMed).
: "Characterization of spike glycoprotein of SARS-CoV-2 on virus entry and its immune cross-reactivity with SARS-CoV." dans: Nature communications, Vol. 11, Issue 1, pp. 1620, (2020) (PubMed).
: "Biochemical characterization of SARS-CoV-2 nucleocapsid protein." dans: Biochemical and biophysical research communications, Vol. 527, Issue 3, pp. 618-623, (2020) (PubMed).
: "A SARS-CoV-2-Human Protein-Protein Interaction Map Reveals Drug Targets and Potential Drug-Repurposing." dans: bioRxiv : the preprint server for biology, (2020) (PubMed).
: "Immunoinformatic analysis of the SARS-CoV-2 envelope protein as a strategy to assess cross-protection against COVID-19." dans: Microbes and infection, Vol. 22, Issue 4-5, pp. 182-187, (2020) (PubMed).
: "Cell entry mechanisms of SARS-CoV-2." dans: Proceedings of the National Academy of Sciences of the United States of America, Vol. 117, Issue 21, pp. 11727-11734, (2020) (PubMed).
- Jie Hu et al. The D614G mutation of SARS-CoV-2 spike protein enhances viral infectivity and decreases neutralization sensitivity to individual convalescent sera. bioRxviv (2020).
- Korber B. et al.Spike mutation pipeline reveals the emergence of a more transmissible form of SARS-CoV-2. bioRxviv (2020). doi.org/10.1101/2020.04.29.069054.
- Lizhou Zhang et al. The D614G mutation in the SARS-CoV-2 spike protein reduces S1 shedding and increases infectivity. bioRxviv (2020). doi.org/10.1101/2020.06.12.148726.
- Junxian Ou et al. Emergence of RBD mutations in circulating SARS-CoV-2 strains enhancing the structural stability and human ACE2 receptor affinity of the spike protein. bioRxiv (2020). doi:10.1101/2020.03.15.991844v4
- Saha, P. et al.Mutations in Spike Protein of SARS-CoV-2 Modulate Receptor Binding, Membrane Fusion and Immunogenicity: An Insight into Viral Tropism and Pathogenesis of COVID-19. chemRxiv (2020). doi:10.26434/chemrxiv.12320567.v1




