CUT&RUN, qui signifie "cleavage under targets and release using nuclease", offre une nouvelle approche de l'épigénétique1.
CUT&RUN surmonte les divers inconvénients du ChIP-Seq grâce à un flux de travail amélioré. CUT&RUN est simple à réaliser et est intrinsèquement robuste, avec un bruit de fond extrêmement faible ne nécessitant qu'un dixième de la profondeur de séquençage de ChIP, ce qui rend CUT&RUN particulièrement rentable pour le profilage des facteurs de transcription et de la chromatine.1.
Cependant, il ne se limite pas au profilage; CUT&RUN a le potentiel de remplacer toutes les applications basées sur le ChIP.
Regardez notre vidéo pour avoir une introduction au sujet CUT&RUN!
CUT&RUN - Combiner les avantages des variations de ChIP-Seq
Comme CUT&RUN est réalisé sur des cellules ou des noyaux intacts sans fragmentation, il peut être utilisé pour sonder tous les compartiments génomiques. Les complexes chromatiniens clivés diffusent hors des noyaux où ils peuvent être récoltés dans le surnageant. Le reste du génome non digéré est conservé dans les noyaux intacts. En revanche, dans le cas de la technique ChIP-seq, la majorité de la chromatine est cisaillée ou digérée, ce qui se traduit, en comparaison, par un rapport signal/bruit moins bon. Par conséquent, le ChIP-Ssq nécessite une profondeur de séquençage plus importante et, à chaque cycle de séquençage, une quantité d'échantillons supplémentaire, du temps de travail et de l'argent.
CUT&RUN Flux de Travail: Extraction sans fragmentation
Le CUT&RUN est simple et peut être réalisé en moins d'une journée en utilisant un équipement de laboratoire standard. CUT&RUN est adapté à des applications allant jusqu'à 100 cellules pour le profilage de H3K27me3 ou 1000 cellules pour la protéine de liaison à l'ADN spécifique à la séquence CTCF.1.
Par conséquent, CUT&RUN permet d'établir des cartes ciblées des interactions protéine-ADN à l'échelle du génome, même pour les types de cellules rares. Par rapport aux données XChiP-seq, les deux séries CUT&RUN révèlent des pics fiables avec moins de bruit de fond. L'un des points forts de CUT&RUN est que la réaction est entièrement réalisée in situ.
Discover our CUT&RUN Antibodies!
Show moreCUT&RUN : Questions fréquemment posées
- Quels sont les avantages et les inconvénients de CUT&RUN et CUT&Tag par rapport à ChIP-seq? How do I choose between both methods.
- Pourquoi le rendement de l'ADN est-il si faible?
- Comment choisir un anticorps primaire pour CUT&RUN ou CUT&Tag?
- Pourquoi ai-je besoin d'un anticorps témoin négatif pour CUT&RUN? Pourquoi ne pas simplement utiliser un contrôle sans anticorps?
- Puis-je remplacer le contrôle négatif de l'anticorps pour CUT&RUN en utilisant un knock-out (ou knock-down) de ma protéine?
- Dois-je utiliser un anticorps secondaire pour CUT&RUN et CUT&Tag?
- L'un de mes anticorps est de type souris. Votre pAG-MNase a-t-elle une bonne affinité pour les anticorps de souris, ou conseillez-vous d'utiliser un anticorps secondaire de lapin anti-souris?
- Dois-je inclure de l'ADN hétérologue pour la quantification?
- Est-il possible de fixer les cellules avant l'immobilisation?
- Est-il possible d'utiliser les ensembles de produits anticorps-online CUT&RUN avec des échantillons de tissus végétaux?
- Est-il possible d'adapter CUT&RUN aux protéines de liaison de l'ARN?
- Au lieu de la digestion par la protéinase K, puis-je dénaturer les protéines des complexes de produits CUT&RUN par la chaleur?
- Qu'est-ce qui est préférable pour les extractions d'ADN avant la préparation de la bibliothèque : l'extraction au phénol-chloroforme ou la purification par affinité à l'aide d'une colonne?
- Est-il possible de faire un séquençage simple au lieu d'un séquençage en paires des bibliothèques CUT&RUN?
Manuel CUT&RUN + CUT&Tag!
Obtenez votre exemplaire gratuitQuels sont les avantages et les inconvénients de CUT&RUN et CUT&Tag par rapport à ChIP-seq? Comment puis-je choisir entre les deux méthodes.
Les avantages de CUT&RUN et CUT&Tag par rapport à ChIP-seq sont un meilleur rapport signal/bruit, une sensibilité plus élevée, une plage dynamique plus large, une exigence moindre en termes de lectures de séquençage et de nombre de cellules.CUT&Tag présente l'avantage que les amorces de séquençage sont fixées aux fragments d'ADN clivés et nécessite moins d'étapes de préparation de bibliothèque que CUT&RUN. Aucun recuit supplémentaire n'est nécessaire. Cette méthode fonctionne particulièrement bien pour les protéines nucléosomiques et les protéines à liaison étroite. Elle a également été rationalisée par le laboratoire Henikoff en un protocole où l'ensemble du processus se déroule dans un seul tube et des variations à haut débit se prêtant à l'automatisation sont disponibles.
CUT&RUN d'autre part est préférable pour les facteurs de transcription et d'autres protéines de liaison à l'ADN moins étroitement liées qui sont sensibles à la concentration de sel plus élevée dans CUT&Tag nécessaire pour empêcher le marquage hors cible de la chromatine accessible par Tn5. En outre, la résolution spatiale de la digestion par la MNase est plus élevée que celle de la tagmentation, ce qui permet d'obtenir une empreinte plus nette de la protéine d'intérêt.
Pourquoi le rendement en ADN est-il si faible?
CUT&RUN et CUT&Tag sont réalisés en utilisant un faible nombre de cellules et le signal de fond est considérablement plus faible que par exemple pour le ChIP. Cela peut rendre difficiles les mesures fiables de la concentration d'ADN à l'aide d'un dosage fluorométrique ou par électrophorèse capillaire.
Pour évaluer le succès des méthodes CUT&RUN et CUT&Tag, il est recommandé d'inclure une réaction utilisant un anticorps contre et une modification d'histone abondante telle que h4K27me ( ABIN6923144) ou h4K4me3 ( ABIN2668472) comme contrôle positif. Les fragments d'ADN préparés à l'aide d'un tel anticorps peuvent être mesurés par électrophorèse capillaire sur un Bioanalyzer ou Tapestation ou par fluorométrie sur un fluoromètre Qubit ou Nanodrop.
Comment choisir un anticorps primaire pour CUT&RUN ou CUT&Tag?
Les anticorps qui sont recommandés pour ChIP-seq ne fonctionnent pas nécessairement dans CUT&RUN dans CUT&Tag. Contrairement à ChIP-seq, l'antigène est généralement dans son état natif sans fixation supplémentaire. À moins qu'un anticorps ait déjà été testé pour CUT&RUN/Tag, il est utile de recommander une méthode dans laquelle l'antigène est censé être à l'état natif, par exemple l'immunofluorescence. Sauf indication contraire, la dilution recommandée pour l'immunofluorescence est également un bon point de départ pour la concentration de l'anticorps en CUT&RUN/Tag.
Pourquoi ai-je besoin d'un anticorps de contrôle négatif pour CUT&RUN? Pourquoi ne pas simplement utiliser un contrôle sans anticorps?
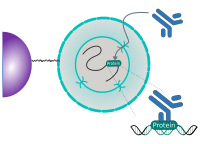
La MNase utilisée pour CUT&RUN est une endo- et exonucléase qui va se lier de manière non spécifique et cliver l'ADN non protégé dans l'ADN hyper-accessible, par exemple dans les régions entourant les éléments régulateurs. La MNase libre coupera préférentiellement l'ADN à l'intérieur de ces régions hyper-accessibles, ce qui peut potentiellement provoquer des faux positifs et augmenter le signal de fond en général.
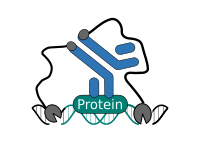
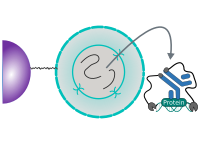
Pour éviter cet effet indésirable de la MNase non attachée, la chromatine est recouverte de manière aléatoire avec l'anticorps de contrôle négatif CUT&RUN guinea pig anti-rabbit IgG (ABIN101961) avant l'ajout de pAG-MNase ( ABIN6950951) aux échantillons. La pAG-MNase est alors attachée via sa partie Protéine A ou Protéine G au fragment Fc de l'anticorps et la fragmentation de l'ADN de fond est dictée par la liaison aléatoire de l'anticorps par opposition à la digestion par nucléase des régions d'ADN hyper-accessibles.
Puis-je remplacer le contrôle négatif de l'anticorps pour CUT&RUN en utilisant un knock-out (ou knock-down) de ma protéine?
Les deux contrôles sont utiles mais portent sur différents aspects de l'expérience et ne sont donc pas interchangeables.
L'anticorps de contrôle négatif CUT&RUN guinea pig anti-rabbit IgG (ABIN101961) est utilisé pour établir un fond de référence pour l'appel des pics. Cela est nécessaire en raison du faible signal de fond dans les échantillons CUT&RUN par rapport aux échantillons ChIP-seq. Le contrôle ko (ou kd), quant à lui, donne une impression de liaison non spécifique de l'anticorps dirigé contre la protéine d'intérêt à d'autres protéines. Il est utile pour éviter l'identification de signaux faussement positifs.
Dois-je utiliser un anticorps secondaire pour CUT&RUN et CUT&Tag?
Selon l'espèce hôte et l'isotype de l'anticorps et de la protéine A et/ou de la protéine G de fusion MNase, un anticorps secondaire peut être nécessaire pour la liaison MNase. La protéine A a une bonne affinité élevée avec tous les anticorps IgG de lapin mais une faible affinité avec les anticorps IgG isotypes de rat, de chèvre et de mouton et certaines sous-classes d'anticorps IgG de souris, en particulier l'IgG1. En revanche, la protéine G se lie bien à la région Fc des IgG de souris, de chèvre, de mouton et de la plupart des IgG de rat. Son affinité pour les IgG de lapin est toutefois inférieure à celle de la protéine A. Lors de l'utilisation de la pAG-MNase introduite avec le protocole CUT&RUN amélioré, il n'est donc généralement pas nécessaire d'utiliser un anticorps secondaire. L'utilisation de la pA-MNase du protocole original pourrait cependant nécessiter l'utilisation d'un anticorps secondaire élevé en lapin pour assurer une liaison efficace de la protéine de fusion à l'anticorps.
Pour CUT&Tag, un anticorps secondaire est recommandé pour augmenter la concentration locale du site de liaison du fragment Fc à proximité du site de transposition prévu autour de l'antigène d'intérêt. Cette étape est nécessaire pour augmenter le signal spécifique.
Un de mes anticorps est une souris. Votre pAG-MNase a-t-elle une bonne affinité pour les anticorps de souris, ou conseillez-vous d'utiliser un anticorps secondaire lapin anti-souris?
La pAG-MNase fonctionnera bien avec votre anticorps murin. L'ajout de la protéine G à la MNase est principalement destiné à s'adapter à l'utilisation d'anticorps monoclonaux IgG1 de souris qui se lient faiblement à la protéine A. Les autres isotypes d'IgG se lient bien soit à la protéine A, soit à la protéine G.
Dois-je inclure de l'ADN spike-in hétérologue pour la quantification?
Notre protocole est largement basé sur le protocole amélioré CUT&RUN. Ici, les auteurs montrent qu'une quantification précise est possible en utilisant de l'ADN spike-in hétérologue ou de l'ADN E. coli reporté de la purification pAG-MNase. Par conséquent, l'ajout d'ADN spike-in hétérologue n'est pas nécessaire.
Est-il possible de fixer les cellules avant l'immobilisation?
Il est possible de fixer vos échantillons, par exemple pour éviter la dissociation d'un complexe protéique plus important de l'ADN au cours de l'expérience. Vous pouvez soit suivre votre procédure de réticulation établie, soit des conditions de réticulation douces en utilisant du formaldéhyde à une concentration plus faible de 0,1%. La réticulation à 1% de formaldéhyde peut en fait réduire le signal, probablement en raison du masquage des épitopes. Dans ces cas, une concentration plus faible de réticulant est préférable.
Est-il possible d'utiliser les ensembles de produits CUT&RUN d'anticorps-online avec des échantillons de tissus végétaux?
La méthode CUT&RUN peut être appliquée à des échantillons de tissus végétaux. Une étape essentielle, en plus de celles indiquées dans le protocole, est la génération de sphéroblastes afin qu'il devienne possible de perméabiliser la membrane plasmique pour l'application des anticorps et de la protéine de fusion MNase. On peut également utiliser des noyaux isolés comme matériel d'échantillonnage.
L'anticorps de contrôle positif CUT&RUN rabbit anti-h4K27me3 ( ABIN6923144) et l'anticorps témoin négatif CUT&RUN guinea pig anti-rabbit IgG (ABIN101961) ainsi que les billes de ConA (ABIN6923139 ou ABIN6952467) sont adaptés à une utilisation avec des échantillons de plantes.
Est-il possible d'adapter CUT&RUN aux protéines de liaison à l'ARN?
La MNase utilisée pour CUT&RUN accepte également l'ARN comme substrat, il serait donc possible d'adapter le protocole CUT&RUN pour l'utiliser sur les protéines de liaison à l'ARN.
L'ARN dans le cytoplasme attirera la machinerie de dégradation s'il est dépourvu de la coiffe 5' et de la queue poly-A 3'. Par conséquent, je suggère d'utiliser le travail avec des noyaux isolés. Cela présente l'avantage supplémentaire de pouvoir omettre la digitonine dans les tampons, qui est utilisée pour remplacer le cholestérol et perméabiliser la membrane cellulaire. Les noyaux isolés peuvent ensuite être immobilisés à l'aide de billes magnétiques ConA comme pour une expérience CUT&RUN. Un anticorps contre la protéine d'intérêt est ajouté et ensuite la pAG-MNase est attachée à l'anticorps, amenant ainsi la MNase à proximité de l'ARN d'intérêt. L'ARN isolé peut ensuite être transcrit de manière inverse en ADNc pour produire votre bibliothèque de séquençage.
Afin d'éliminer tout ADN contaminant, incluez un traitement à la DNase dans le protocole après la préparation de l'ARN et avant la préparation de la bibliothèque. La normalisation basée sur l'ADN E. coli emporté avec la MNase ou un ADN spike-in n'est pas une option dans ce cas. Envisagez plutôt d'utiliser le nombre total de lectures pour normaliser entre les échantillons ou d'inclure un ARN de référence d'une concentration connue.
Au lieu de la digestion à la protéinase K, puis-je dénaturer les protéines des complexes de produits CUT&RUN par la chaleur?
Nous déconseillons cette option : l'ADN d'intérêt est à son moment présent dans un complexe constitué de l'ADN, de l'antigène, de l'anticorps correspondant et de la pAG-MNase. L'ébullition de ce complexe précipitera probablement l'ADN avec la protéine dénaturée. Cela affectera aussi principalement les produits CUT&RUN courts et non les molécules d'ADN plus grandes, ce qui entraînera une diminution du rapport signal/bruit dans votre bibliothèque et réduira potentiellement aussi la complexité de la bibliothèque. Cet effet est encore exacerbé en raison de la température de fusion plus basse de ces molécules courtes par rapport aux molécules d'ADN contaminantes plus longues.
Qu'est-ce qui est préférable pour les extractions d'ADN avant la préparation de la bibliothèque : l'extraction par phénol-chloroforme ou la purification par affinité à l'aide d'une colonne?
Un problème potentiel lors de l'utilisation de billes SPRI pour le nettoyage du fragment d'ADN est le report de la protéinase K active, qui peut interférer avec l'amplification PCR en aval. Par conséquent, une extraction phénol-chloroforme est préférable pour assurer une dénaturation complète de la protéinase K.
Est-il possible d'effectuer un séquençage à simple extrémité au lieu d'un séquençage en paires des bibliothèques CUT&RUN?
Le séquençage single-end au lieu du séquençage paired-end est possible. Cependant, il présente des inconvénients par rapport au séquençage en paires : (i) Pour les cibles abondantes comme les marques d'histones ou les facteurs de transcription, un grand nombre de sites de liaison est attendu. Le séquençage en paires facilite le mappage sans ambiguïté à la position génomique correcte. Ces informations supplémentaires réduisent la profondeur de séquençage nécessaire. (ii) La MNase digère l'ADN cible jusqu'à la section couverte par la protéine d'intérêt. Le séquençage pair-end révélera cette empreinte alors que l'information est perdue dans le séquençage single-end.
Conseils pour la sélection des anticorps
La réussite de l'exécution du clivage sous cible et de la libération à l'aide d'une nucléase (CUT&RUN) repose sur la sélection des anticorps. Un anticorps spécifique d'un facteur ou d'une histone est lié à la chromatine in situ, suivi de la liaison à l'anticorps d'une fusion protéine A-nucléase micrococcique (pA-MNase). Comme c'est le cas pour la ChIP, le succès dépend en grande partie de l'affinité de l'anticorps pour sa cible et de sa spécificité dans les conditions utilisées pour la liaison. Nous avons compilé ci-dessous une liste d'anticorps primaires et secondaires adaptés au CUT&RUN. En outre, antibodies-online soutient la validation des anticorps pour CUT&RUN. Vous pouvez participer à notre independent validation initiative (IVI). Proposez et réalisez une expérience de validation et obtenez un remboursement complet de l'anticorps validé. Veuillez nous contacter, si vous êtes intéressé par la validation de l'un de nos produits CUT&RUN !
.Quels anticorps secondaires puis-je utiliser pour CUT&RUN?
- (201)
- (3)
- (2)
Quels anticorps primaires puis-je utiliser pour CUT&RUN?
- (11)
- (14)
- (1)
- (12)
- (15)
- (1)
- (6)
- (21)
- (1)
- (2)
- (4)
- (1)
- (1)
- (4)
- (1)
- (24)
- (5)
- (1)
- (3)
- (5)
- (1)
- (4)
- (5)
- (1)
- (1)
- (2)
- (1)
- (1)
- (2)
- (1)
- (75)
- (5)
- (2)
- (1)
- (2)
- (1)
- (2)
- (1)
- (5)
- (10)
- (8)
- (2)
- (1)
- (1)
- (5)
- (5)
- (2)
- (5)
Références:
- Peter J. Skene and Steven Henikoff (2018): "CUT&RUN: Targeted in situ genome-wide profiling with high efficiency for low cell numbers”. Nature, Volume 13, pages 1006–1019. [PMID: 25652980]
- Sandipan Brahma and Steven Henikoff (2018): "RSC-Associated Subnucleosomes Define MNase-Sensitive Promoters in Yeast". Mol Cell, Volume 73, Issue2, P238-249. [DOI]
KEYWORDS:
Cut and Run, CutAndRun, ChIP-seq, ChIP-sequencing, chromatin; chromosomes; epigenomics; gene expression; genetics; genomics; human; spike-in calibration; in situ profiling; transcription factors; A-Micrococcal Nuclease

Creative mind of antibodies-online with a keen eye for details. Proficient in the field of life-science with a passion for plant biotechnology and clinical study design. Responsible for illustrated and written content at antibodies-online as well as supervision of the antibodies-online scholarship program.
Aller à la page de l’auteur




