L'effet Warburg
Les altérations oncogènes du métabolisme cellulaire ont longtemps été considérées comme un effet secondaire du cancer, précipité par des modifications génétiques. Plus récemment, cette perception a changé et la dérégulation de l'énergétique cellulaire est désormais comprise dans les Hallmarks of Cancer depuis leur deuxième itération en 2011. Cette perception est corroborée par le fait que de nombreuses mutations cancéreuses sont également impliquées dans le métabolisme cellulaire et que l'on estime que deux tiers des cancers présentent des mutations dans les gènes glycolytiques.
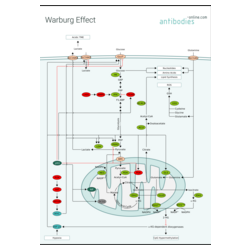
L'adaptation la plus connue du métabolisme des cellules cancéreuses est l'effet Warburg, ou glycolyse aérobie. Il doit son nom à Otto Heinrich Warburg qui a observé dans les années 1920 la production d'acide lactique dans les cellules de tumeurs dans des conditions aérobies. L'effet Warburg décrit la préférence des tumeurs pour la fermentation du glucose en lactate, même en présence de quantités suffisantes d'oxygène. Le pyruvate, produit final de la glycolyse, est réduit en lactate au lieu d'être transporté dans les mitochondries pour la phosphorylation oxydative par le cycle de l'acide citrique. Le lactate est transporté hors de la cellule et contribue à l'acidification du microenvironnement tumoral (TME).
L'efficacité catabolique de la glycolyse aérobie est considérablement plus faible que celle de la phosphorylation oxydative : la glycolyse aérobie ne produit que 2 molécules d'ATP par molécule de glucose alors que la phosphorylation oxydative produit entre 32 et 34 molécules d'ATP par molécule de glucose. Cependant, elle procure un avantage sélectif différent aux cellules cancéreuses en fournissant les éléments essentiels - nucléotides, acides aminés et lipides - nécessaires à la prolifération rapide des cellules cancéreuses. En raison du détournement du pyruvate vers le lactate, le glutamate devient la source principale de carbone pour reconstituer les intermédiaires métaboliques du cycle l'acide citrique dans les mitochondries afin de couvrir les besoins énergétiques des cellules cancéreuses.Des mutations de gain de fonction dans l'isocitrate déshydrogénase dans le cytosol (IDH1) et dans les mitochondries (IDH2) entraînent la réduction du α-cétoglutarate (α-KG) - l'un des métabolites du cycle de l'acide citrique - en R-2-hydroxyglutarate (2-HG). Cet oncométabolite inhibe les dioxygénases dépendantes de α-KG en diminuant la concentration de leur cofacteur obligatoire α-KG. La classe des dioxygénases dépendantes de α-KG comprend des déméthylases diverses et méthyltransférases modifiant la chromatine. Leur inhibition entraîne une hyperméthylation des îlots CpG et affecte le destin cellulaire. Les dioxygénases dépendantes de α-KG comprennent également des prolyl hydroxylases, qui influencent l'activité du facteur 1 inductible par l'hypoxie (HIF1), un régulateur principal de la transcription dans la réponse adaptative à l'hypoxie.
Il a été démontré que la régulation de facteurs de transcription comme HIF1 et Myc et l'activation de voies de signalisation comme la signalisation PI3K/AKT contribuent au phénotype de Warburg dans le cancer. L'inactivation de suppresseurs de tumeurs comme p53 est également un mécanisme important. Dans des conditions normales, p53 régule négativement la glycolyse et favorise la phosphorylation oxydative. Cependant, ces mécanismes ne fonctionnent pas dans des conditions de glycolyse aérobie, ce qui favorise la croissance continue et la survie des cellules cancéreuses.
Voies de signalisation liées :
Références bibliographiques :
- : "Regulation of cancer cell metabolism." dans: Nature reviews. Cancer, Vol. 11, Issue 2, pp. 85-95, (2011) (PubMed).
- : "Hallmarks of cancer: the next generation." dans: Cell, Vol. 144, Issue 5, pp. 646-74, (2011) (PubMed).
- : "Glutamine oxidation maintains the TCA cycle and cell survival during impaired mitochondrial pyruvate transport." dans: Molecular cell, Vol. 56, Issue 3, pp. 414-24, (2014) (PubMed).
- : "Glucose transporters in cancer - from tumor cells to the tumor microenvironment." dans: The FEBS journal, Vol. 285, Issue 16, pp. 2926-2943, (2019) (PubMed).
- : "Warburg and Krebs and related effects in cancer." dans: Expert reviews in molecular medicine, Vol. 21, pp. e4, (2020) (PubMed).
- : "Targeting Glucose Transporters for Breast Cancer Therapy: The Effect of Natural and Synthetic Compounds." dans: Cancers, Vol. 12, Issue 1, (2020) (PubMed).
- : "Metabolic Coordination of Cell Fate by α-Ketoglutarate-Dependent Dioxygenases." dans: Trends in cell biology, Vol. 31, Issue 1, pp. 24-36, (2021) (PubMed).
- : "Regulation of Glycolysis by Non-coding RNAs in Cancer: Switching on the Warburg Effect." dans: Molecular therapy oncolytics, Vol. 19, pp. 218-239, (2020) (PubMed).
- : "Hallmarks of Cancer: New Dimensions." dans: Cancer discovery, Vol. 12, Issue 1, pp. 31-46, (2022) (PubMed).